什么是SPEI 标准化降水蒸散指数SPEI(Standardized Precipitation Evapotranspiration Index)可以表征干湿状态。提出 。
SPEI算法 注意 :这里的SPEI计算方法存在一些缺陷。目前已经基于其他包并行化之后的算法对其进行修改,请等待博客后续更新。
构建差值序列 首先构建降水量$P_i$和潜在蒸散发$PET_i$的差值序列$D_i$,
$$ D_i = P_i-PET_i $$
可以选定不同的时间尺度来计算SPEI。时间尺度为$k$,$i$日的$D_i$累计为该日前$k$天的$D_i$之和,也就是说,对于每个$k$尺度的$D_i$,其为前$k$天的滑动窗口和。
对差值序列进行拟合 目前常见的SPEI拟合方法均为使用三参数Log-Logistic分布(也称Fisk分布)对$D_i$进行拟合。近几年的研究显示,Genextreme分布对SPEI有更好的拟合效果 。
Python在GIS系统中有其重要的生态位,但是使用Python对SPEI进行计算十分低效。传统方法中,若使用Scipy和循环进行Fisk分布的参数估计,计算大型数据集将十分困难,考虑如下代码:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 import numpy as npdef calc_spei_fisk_paras (d_i_matrix:np.ndarray ) -> np.ndarray: from scipy.stats import fisk d_i_shape = d_i_matrix.shape paras = np.zeros_like(d_i_shape)[0 :3 ,:,:] for i in d_i_shape[1 ]: for j in d_i_shape[2 ]: paras[:,i,j] = fisk.fit(d_i_matrix[:,i,j]) return paras
该函数使用二重嵌套循环对SPEI进行计算,因为Python的特性,该段代码的执行将极度缓慢,面对高分辨率图象时尤其明显。要对该算法进行优化,有以下几个方向:
概率值正态化 序列值代入已估计参数的Genextreme分布得到概率值$P_i$;之后将概率值代入标准正态分布累积分布函数的反函数,得到SPEI值:
$$ \mathrm{SPEI}_i = \Phi^{-1}(P_i) $$
算法优化 循环向量化 循环向量化指将使用循环进行的操作改为操作向量,C语言在编译时编译器会自动地将循环优化成处理器支持的SIMD指令。
SIMD(Single Instruction Multiple Data)即单指令流多数据流,是一种采用一个控制器来控制多个处理器,同时对一组数据(又称“数据向量”)中的每一个分别执行相同的操作从而实现空间上的并行性的技术。
考虑以下C代码,
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 #include <stdio.h> #define LENGTH(array) (sizeof(array) / sizeof(array[0])) int main () { int vec1[3 ]; int vec2[3 ]; printf ("Input the First Vec:\n" ); scanf ("%d%d%d" , &vec1[0 ], &vec1[1 ], &vec1[2 ]); printf ("Input the Secound Vec:\n" ); scanf ("%d%d%d" , &vec2[0 ], &vec2[1 ], &vec2[2 ]); int vec3[3 ]; for (int i = 0 ; i < LENGTH(vec1); i++) { vec3[i] = vec1[i] + vec2[i]; } printf ("Sum Vector is %d %d %d\n" , vec3[0 ], vec3[1 ], vec3[2 ]); return 0 ; }
该程序将两个int类型的变量使用循环相加。在AMD64机器上使用gcc的-O1和-O3优化选项分别编译得到汇编文件(也可以去网站Compiler Explorer ):
1 2 3 gcc simd.c -S -o simd_O1.S -masm=intel -march=core-avx2 -O1 -v gcc simd.c -S -o simd_O3.S -masm=intel -march=core-avx2 -O3 -v
汇编文件分别为:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 # simd_O1. S .LC0: .string "Input the First Vec:" .LC1: .string "%d%d%d" .LC2: .string "Input the Secound Vec:" .LC3: .string "Sum Vector is %d %d %d\n" main: sub rsp , 40 mov edi , OFFSET FLAT:.LC0 call puts lea rsi , [rsp +20 ] lea rcx , [rsp +28 ] lea rdx , [rsp +24 ] mov edi , OFFSET FLAT:.LC1 mov eax , 0 call __isoc99_scanf mov edi , OFFSET FLAT:.LC2 call puts lea rsi , [rsp +8 ] lea rcx , [rsp +16 ] lea rdx , [rsp +12 ] mov edi , OFFSET FLAT:.LC1 mov eax , 0 call __isoc99_scanf mov ecx , DWORD PTR [rsp +16 ] add ecx , DWORD PTR [rsp +28 ] mov edx , DWORD PTR [rsp +12 ] add edx , DWORD PTR [rsp +24 ] mov esi , DWORD PTR [rsp +8 ] add esi , DWORD PTR [rsp +20 ] mov edi , OFFSET FLAT:.LC3 mov eax , 0 call printf mov eax , 0 add rsp , 40 ret
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 # simd_O3. S .LC0: .string "Input the First Vec:" .LC1: .string "%d%d%d" .LC2: .string "Input the Secound Vec:" .LC3: .string "Sum Vector is %d %d %d\n" main: push rbp mov edi , OFFSET FLAT:.LC0 mov rbp , rsp and rsp , -32 sub rsp , 64 call puts mov rsi , rsp lea rcx , [rsp +8 ] xor eax , eax lea rdx , [rsp +4 ] mov edi , OFFSET FLAT:.LC1 call __isoc99_scanf mov edi , OFFSET FLAT:.LC2 call puts lea rsi , [rsp +32 ] lea rcx , [rsp +40 ] xor eax , eax lea rdx , [rsp +36 ] mov edi , OFFSET FLAT:.LC1 call __isoc99_scanf vmovq xmm0 , QWORD PTR [rsp ] mov ecx , DWORD PTR [rsp +40 ] xor eax , eax vmovq xmm1 , QWORD PTR [rsp +32 ] add ecx , DWORD PTR [rsp +8 ] mov edi , OFFSET FLAT:.LC3 vpaddd xmm0 , xmm0 , xmm1 vpextrd edx , xmm0 , 1 vmovd esi , xmm0 call printf xor eax , eax leave ret
在没有使用优化的情况下,程序使用了3条addl指令,而优化过后其使用了xmm寄存器(SSE指令集)提升了计算效率。但Python解释器没有内建的SIMD支持,要解决这种问题可以使用numpy包:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 import numpy as npimport timeitdef sum_numpy (a:np.ndarray, b:np.ndarray ) -> np.ndarray: return np.sum (a + b) def sum_circulate (a:np.ndarray, b:np.ndarray ) -> np.ndarray: n = len (a) c = np.zeros(n) for i in range (n): c[i] = a[i] + b[i] return np.sum (c) a = np.random.rand(10000 ) b = np.random.rand(10000 ) print (timeit.timeit(lambda : sum_numpy(a, b), number=100 ))print (timeit.timeit(lambda : sum_circulate(a, b), number=100 ))
输出结果不难预测,
1 2 0.004135900000619586 0.6949793999992835
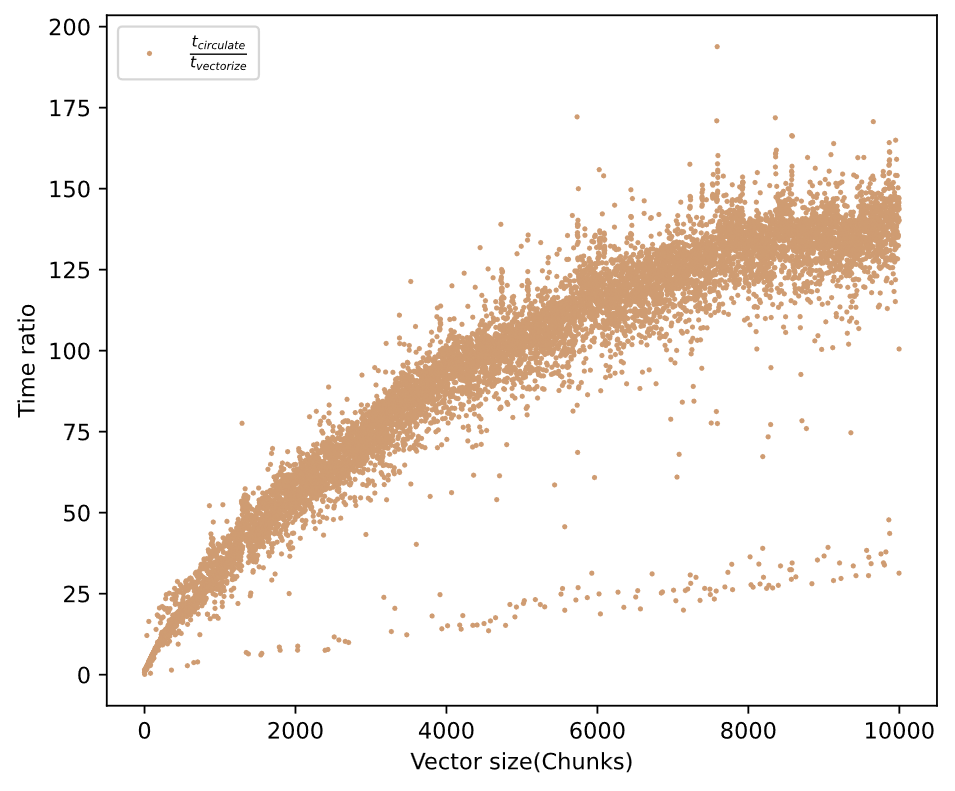
numpy版本的向量加法比循环版本快了170多倍。如果我们以数组规模和所用时间作图,我们得到以下结果:
问题规模越大,SIMD带来的加速效果越明显。但是scipy中的参数估计方法无法实现循环向量化,一次只能处理一个时间序列;而且修改scipy中的代码的成本又太高(大部分代码均由C语言写成),所以需要找新的参数估计模式。
Genextreme分布与L-moments Genextreme分布是相对更好的SPEI拟合的候选分布 。其参数估计可以简单的使用L-moments估计得到 。
使用L-moments的参数估计过程可以由以下公式给出:
GEV分布的CDF为
\begin{equation}
其中$\alpha,\xi,\kappa$为参数。
记L-mean,L-scale,L-skewness分别为$\lambda_1,\lambda_2,\tau_3$,GEV分布的三个参数可以用如下公式近似估计:
\begin{equation}
\begin{equation}
\begin{equation}
线性矩$\lambda_1,\lambda_2,\tau_3$可以使用lmoments3包中的代码修改而来。lmoments3包中的线性矩计算方法可以简单的进行修改,以实现循环向量化:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 import numpy as npimport numba as nb ... @nb.njit(nb.float64[:, :, :](nb.float64[:, :, :], nb.int8 ), parallel=True , cache=True ) def get_lratios_jit (x: np.ndarray, nmom: int = 5 ) -> np.ndarray: """ Calculates L-moments (up to the specified order) of an array of data using Numba JIT compilation. Args: x (np.ndarray): A 3D array of data. nmom (int, optional): The order of L-moments to compute (default is 5). Returns: np.ndarray: An array containing L-moments up to the specified order. """ def sort_array_with_axis (x: np.ndarray, axis: int ) -> np.ndarray: """ Sorts the input array along the specified axis. Args: x (np.ndarray): Input array. axis (int): Axis along which to sort. Returns: np.ndarray: Sorted array. """ for i in nb.prange(x.shape[axis]): x[i] = np.sort(x[i]) return x x = np.asarray(x, dtype=np.float64) n = x.shape[0 ] x = sort_array_with_axis(x, 0 ) sum_xtrans = np.empty_like(x[0 ]) def comb (n, r ): """ Computes the binomial coefficient (n choose r). Args: n (int): Total number of items. r (int): Number of items to choose. Returns: int: Binomial coefficient. """ if r < 0 or r > n: return 0 if r == 0 or r == n: return 1 c = 1 for i in range (1 , r + 1 ): c = c * (n - i + 1 ) // i return c l1 = np.sum (x, axis=0 ) / comb(n, 1 ) if nmom == 1 : return np.expand_dims(l1, axis=0 ) comb1 = np.arange(n) coefl2 = 0.5 / comb(n, 2 ) sum_xtrans[:] = 0 for i in nb.prange(n): sum_xtrans += (comb1[i] - comb1[n - 1 - i]) * x[i] l2 = coefl2 * sum_xtrans if nmom == 2 : return np.stack((l1, l2)) comb3 = np.zeros(n) for i in range (n): comb3[i] = comb(i, 2 ) coefl3 = 1.0 / 3.0 / comb(n, 3 ) sum_xtrans[:] = 0 for i in nb.prange(n): sum_xtrans += ( comb3[i] - 2 * comb1[i] * comb1[n - 1 - i] + comb3[n - 1 - i] ) * x[i] l3 = coefl3 * sum_xtrans / l2 if nmom == 3 : return np.stack((l1, l2, l3)) ...
由以上示例代码能计算出$\lambda_1,\lambda_2,\tau_3$, 便可以由参数估计的公式给出参数值,进而描述Genextreme分布
jit编译 jit 的全称是 Just-in-time,在我们使用的numba库里面则特指Just-in-time Compilation(即时编译)。因为Python的动态类型带来的低效率,需要使用jit技术加速python代码的运行。在代码第一次被执行时将被编译为高效的机器码,同时也能由编译器执行相应的编译优化,以提高执行效率。但是,numba并不是对所有的Python代码都有优化效果,使用前需要去numba的官方文档 查看他支持的python功能和numpy功能。
在上一节中给出的代码就包含了numba包,
1 2 3 4 5 6 import numpy as npimport numba as nb ... @nb.njit(nb.float64[:, :, :](nb.float64[:, :, :], nb.int8 ), parallel=True , cache=True ) def get_lratios_jit (x: np.ndarray, nmom: int = 5 ) -> np.ndarray: ...
注意到使用了@njit函数装饰器,装饰器的参数里给出了输入输出数组的数据类型,以及parallel=True,cache=True等几个选项。他们的意义分别是开启自动循环并行化和编译缓存。这里将仅解释循环并行化,也可以看官方对循环自动并行化的解释 。
循环并行化 假设代码在一个多CPU系统上运行,对于类似下文给出的没有交叉迭代以来关系的循环,
1 2 3 4 5 6 7 8 9 10 @njit(parallel=True ) def test (x ): n = x.shape[0 ] a = np.sin(x) b = np.cos(a * a) acc = 0 for i in prange(n - 2 ): for j in prange(n - 1 ): acc += b[i] + b[j + 1 ] return acc
numba会自动将循环分配到CPU的多个核心上以增加并行度。
后记 文中的完整代码已放于Github仓库pylm-spei 。要在正态化的步骤执行加速,可以参考numba-stats 。